i3D-Markers
Mit Hilfe von "i3D-Markers" – wollen Dr. Hayder Amin und Dr. Caghan Kizil vom DZNE-Standort Dresden die Entwicklung von Medikamenten gegen Hirnerkrankungen beschleunigen. Dafür verbinden sie hochdichte Mikroelektroden-Arrays und dreidimensionale Netzwerke menschlicher Nervenzellen zu einem Messsystem. Auf dieses werden zu prüfende Substanzen getropft und mittels künstlicher Intelligenz die Reaktion der Nervenzellen ermittelt. Mit ihrem Verfahren wollen sie die Auswahl aussichtsreicher Wirkstoffkandidaten für klinische Studien optimieren und dazu beitragen, Sackgassen in der Entwicklungspipeline zu vermeiden.
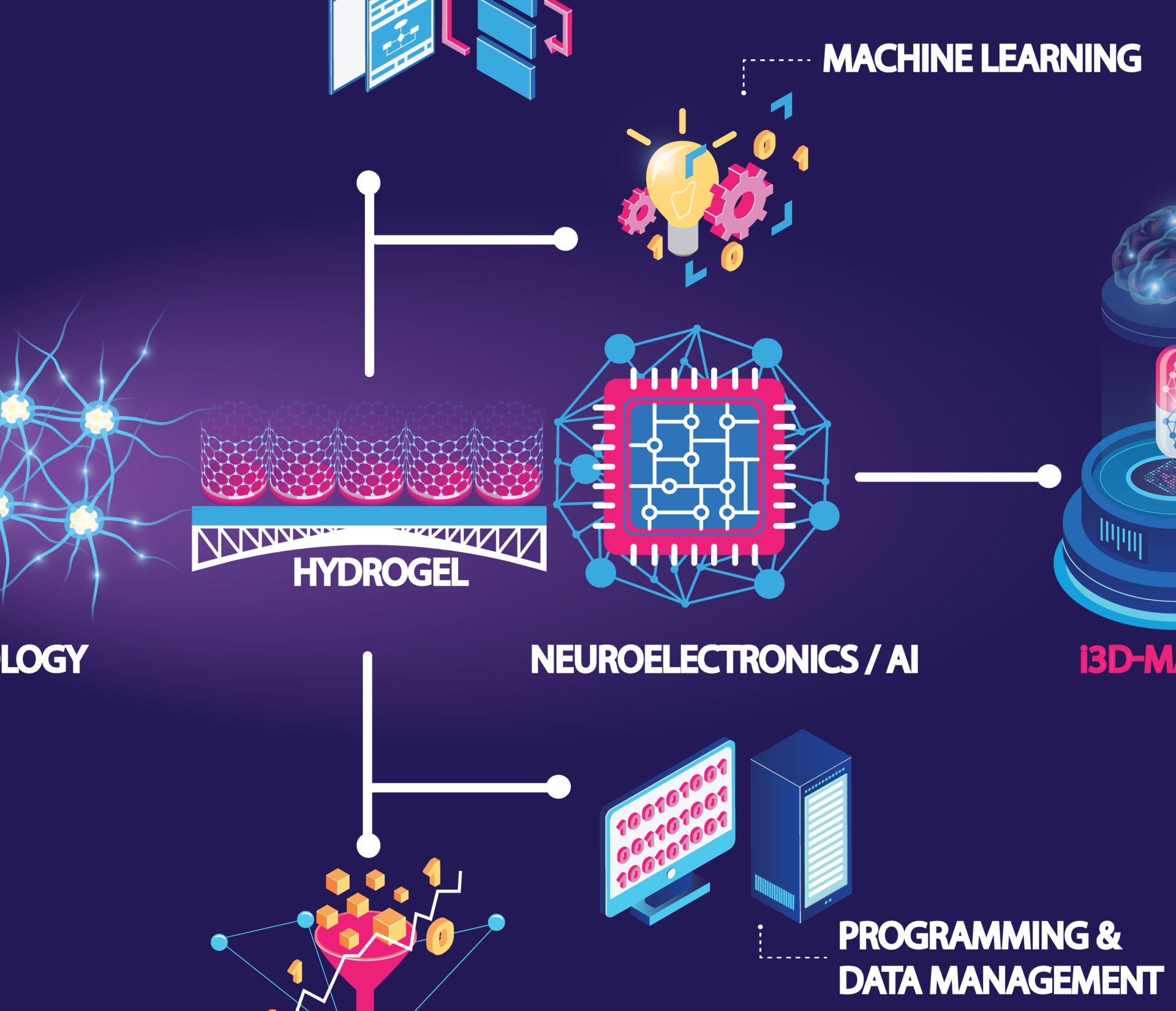
Das Konzept der Dresdner Forscher verschmilzt Neuroelektronik mit einer innovativen Zellkulturmethode. i3D-Markers nutzt neuronale Zellkulturen, die mit einem Mikrochip gekoppelt sind. Das heißt, man züchtet Nervenzellen auf elektronischen Bauteilen, die dicht angeordnete Mikroelektroden beinhalten. Dabei bilden die Nervenzellen ein dreidimensionales Geflecht, wie es im Gehirn vorkommt. Dieser Aufbau ermöglicht es die elektrische Aktivität von Tausenden von Nervenzellen gleichzeitig mit hoher räumlicher und zeitlicher Auflösung zu erfassen. Diese Anordnung von vielen winzig kleinen Sensoren gibt Einblicke in die Dynamik dieses sehr komplexen Netzwerks aus menschlichen Nervenzellen. Damit wird es möglich High-Content-Daten in noch nie dagewesener Detailtiefe und Einzelzellauflösung zu erhalten.
Ein zu testender Wirkstoff wird in definierter Konzentration auf die Nervenzellen getropft. Dann beginnt die Aufzeichnung elektrophysiologischer Daten, die vom Array von Sensoren erfasst werden. Obwohl das Netzwerk aus mehr als hunderttausend Nervenzellen besteht, würde diese Zellkultur einschließlich des Mikrochips auf eine Fläche passen, die zehnmal kleiner ist als eine 2-Eurocent-Münze. Die Informationen über die neuronale Aktivität werden allerdings komplex sein, da sie von Tausenden von Mikrokontaktsensoren stammen. Für die Analyse werden daher Methoden der künstlichen Intelligenz eingesetzt. Diese Algorithmen identifizieren und extrahieren Muster aus komplexen, mehrdimensionalen Daten. Durch die künstliche Intelligenz und spezielle mathematische Ansätze werden sogenannte funktionelle Biomarker entwickelt, die auf der Aktivität des neuronalen Netzwerks beruhen. Diese Biomarker werden mitteilen, ob es dem Netzwerk gut geht oder nicht und ob ein bestimmter Wirkstoff diesen Zustand beeinflusst. Das wird helfen, vielversprechende Wirkstoffe zu identifizieren.
Die Validierung dieses Ansatzes wird mit 820.000 Euro aus dem „Helmholtz-Validierungsfonds“ und mit Mitteln des DZNE finanziert, so dass sich das Gesamtbudget auf rund 1,2 Millionen Euro beläuft. Ziel ist es, die neue Technologie für den industriellen Einsatz fit zu machen.
Projektleiter