Forschungsschwerpunkt
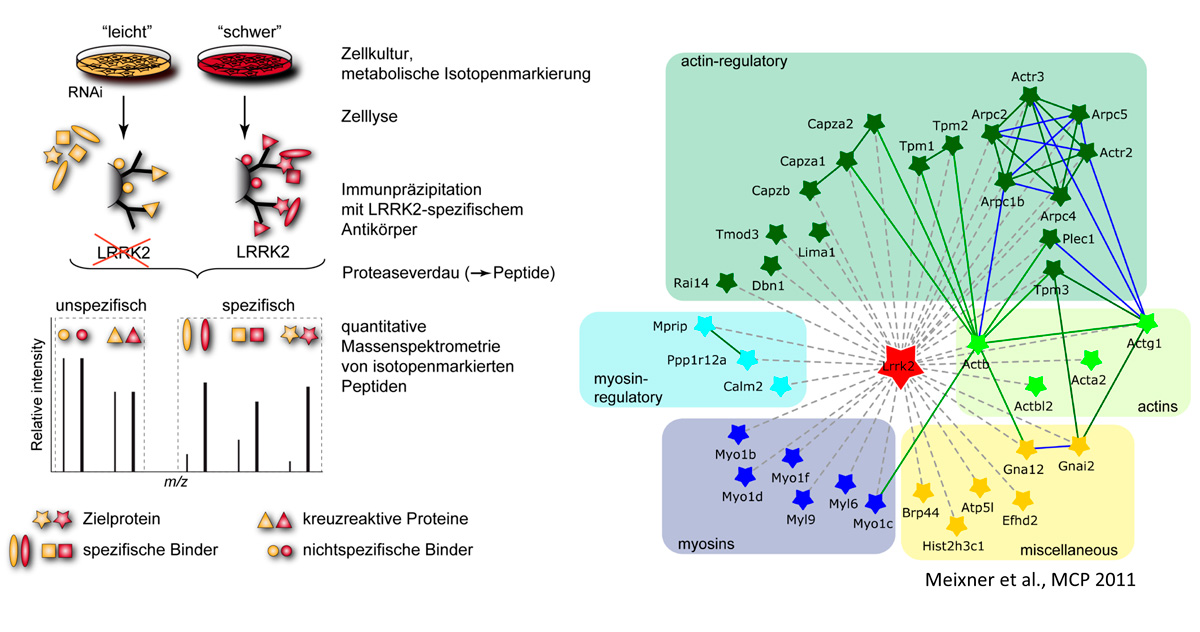
Die Arbeitsgruppe beschäftigt sich mit der Analyse funktioneller Proteinnetzwerke von Proteinen, die mit neurodegenerativen Erkrankungen, speziell Morbus Parkinson, assoziiert sind. Im Sinne eines ‚Guilt-by-Association‘ (Schuld durch Zugehörigkeit)-Prinzips werden mutationsspezifische Veränderungen funktioneller Proteinnetzwerke bestimmt, um die, dem erblich bedingten Morbus Parkinson zu Grunde liegenden, molekularen Pathomechanismen aufzuklären. Dabei wird eine affinitätsbasierte Aufreinigung mit verschiedenen Methoden der quantitativen Massenspektrometrie kombiniert, um Protein-Protein-Interaktionsnetzwerke krankheitsassoziierter Proteine zu identifizieren. Zusätzlich werden komplementäre Methoden zur Identifizierung funktionaler Proteininteraktionen angewandt, z. B. BioID, das auf dem Proximity Labelling Prinzip beruht.
Die Massenspektrometrie wird zusätzlich genutzt, um mutationsspezifische Muster posttranslationaler Modifikationen (z. B. Phosphorylierungen, Acetylierungen oder Ubiquitinylierungen) zu identifizieren.
In Zusammenarbeit mit anderen Arbeitsgruppen des multidisziplinären DZNE-Umfeldes werden die gefundenen Netzwerke nachfolgend in geeigneten Zellkultursystemen, wie patientenderivierten induzierten pluripotenten Stammzellen (iPS Zellen), funktionell validiert.
Die Erkenntnisse aus der systematischen Analyse von mutationsspezifischen Veränderungen von funktionellen Proteinnetzwerken und Signaturen posttranslationaler Modifikationen werden dazu genutzt, hypothesengetrieben Biomarker für Diagnostik und Wirkstofffindung zu identifizieren.
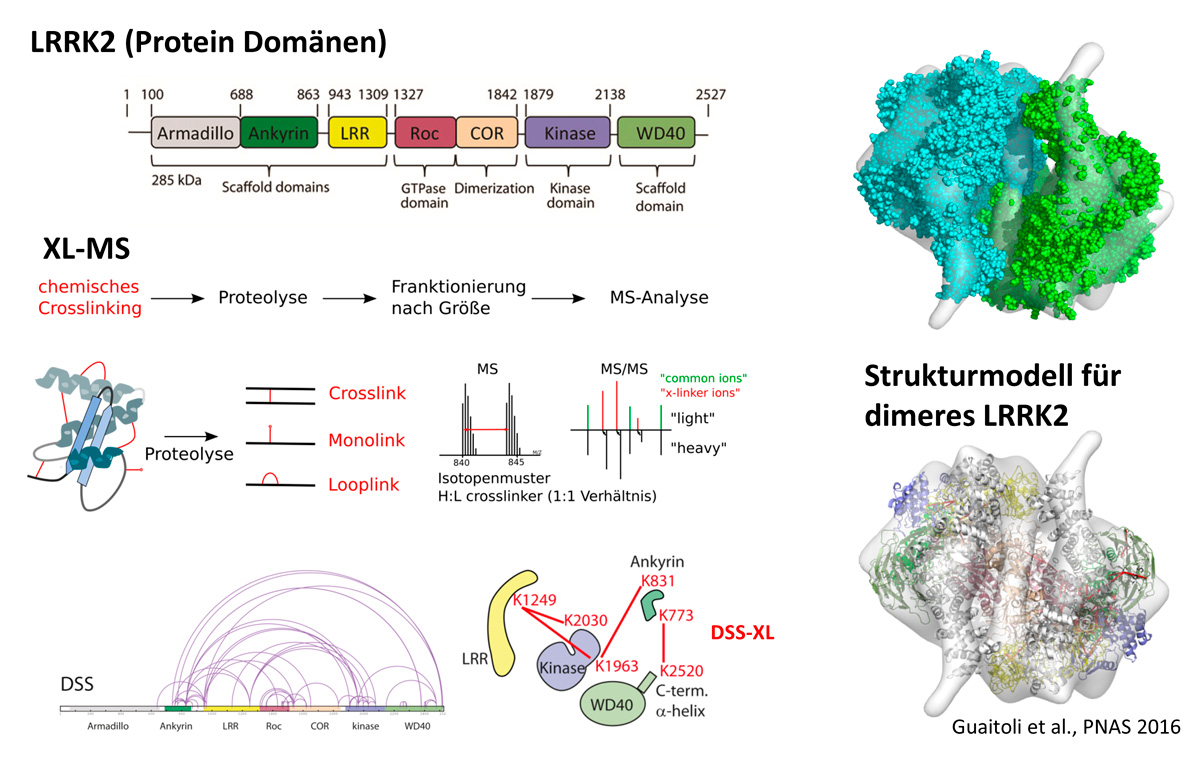
Im Zentrum der Arbeiten steht dabei die systematische Erforschung der Leucine-rich Repeat Kinase 2 (LRRK2), einer Proteinkinase, deren pathogene Mutationen den größten Anteil an erblich bedingtem Morbus Parkinson mit bekannter genetischer Ursache darstellen. Darüber hinaus ist LRRK2 ein vielversprechendes Zielmolekül für eine medikamentöse Therapie, da pathogene Proteinvarianten eine erhöhte Kinaseaktivität bei gleichzeitig verringerter GTPase Aktivität zeigen.
Um die molekularen Mechanismen der intramolekularen Regulation aufzuklären, sowie eine zielgerichtetes Design von Wirkstoffen zu ermöglichen, sind Kenntnisse der Proteinstruktur von zentraler Bedeutung. Das weitverbreitete und auflösungsstarke Verfahren der Röntgenstrukturanalyse lässt sich allerdings auf LRRK2 nur schwer anwenden, da große und flexible Proteine kaum zu kristallisieren sind. Um dennoch eine Vorstellung der räumlichen Struktur von LRRK2 zu bekommen, haben wir einen computergestützten Modellierungsansatz gewählt, in den verschiedene Experimentelle Daten einflossen. Das so erhaltene Strukturmodell von dimerem LRRK2 zeigt eine kompakte Faltung mit Interaktionen von zum Teil weit auseinanderliegenden Proteindomänen welche Teil eines intramolekularen Autoregulationsmechanisms von LRRK2 sein könnten (Guaitoli et al., 2016). Aktuelle Arbeiten unserer Gruppe ziehen nun darauf ab, über eine Verfeinerung des Modells und eingehender biochemischer Charakterisierung die zugrundeliegenden intramolekularen Regulationsmechanismen zu identifizieren.
Förderung:
- Nachwuchsgruppenförderung im Rahmen des iMED Programms der Helmholtz Gemeinschaft
- From structure and function to allosteric targeting of LRRK2-mediated PD; Michael J Fox Foundation LRRK2 Biology (2018)



