München, 7. Februar 2013. Bei manchen Demenzerkrankungen werden stille Bereiche des Erbguts irrtümlich in ungewöhnliche Eiweißstoffe übersetzt. Ein internationales Forscherteam unter Beteiligung des Deutschen Zentrums für Neurodegenerative Erkrankungen (DZNE) in München und der Ludwig-Maximilians-Universität München (LMU) präsentiert diesen Befund in der aktuellen Online-Ausgabe der Fachzeitschrift „Science“. Die nun identifizierten Proteine sollten eigentlich nicht existieren, aber sie bilden den Kern bisher rätselhafter Ablagerungen, die bei genetisch bedingten Störungen des Nervensystems aus dem Spektrum der FTD/ALS-Erkrankungen auftreten. Die Eiweißpartikel sind vermutlich schädlich und könnten daher ein Ansatzpunkt für therapeutische Maßnahmen sein.
Übersetzungsfehler im Gehirn von Demenzpatienten
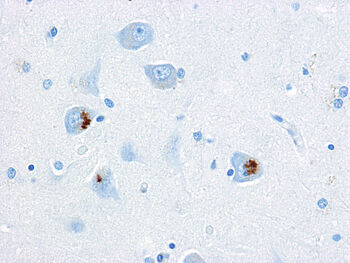
Die Frontotemporale Demenz (FTD), mitunter auch Frontotemporale Lobärdegeneration (FTLD) genannt, und die Amyotrophe Lateralsklerose (ALS) zählen zu einer Gruppe von Erkrankungen des Nervensystems mit überlappenden Symptomen: Die Patienten leiden häufig unter einer Demenz, Persönlichkeitsveränderungen oder auch unter Sprach- und Bewegungsstörungen. Die Krankheit bricht oft schon deutlich vor dem 65. Lebensjahr aus. Die Krankheitsursache ist meist unbekannt, in etwa 30 Prozent der Fälle ist sie genetisch bedingt. In Europa zeigt sich bei etwa 10 Prozent aller erkrankten Personen ein besonderer Gendefekt: In der DNA – dem Träger der Erbinformation – ist ein bestimmter Ausschnitt in überlanger Kopie aneinandergereiht. Gleichzeitig finden sich in den Hirnzellen dieser Patientengruppe Eiweißpartikel, deren Zusammensetzung bislang unbekannt war. Das Team um die Münchner Molekularbiologen Dieter Edbauer und Christian Haass konnte jetzt nachweisen, dass beide Beobachtungen – Gendefekt und Eiweißpartikel - miteinander in Verbindung stehen.
„Wir haben festgestellt, dass diese Proteine auf eine genetische Besonderheit zurückgehen, die bei vielen Patienten auftritt. An einer Stelle im Gen C9orf72 finden sich hundertfache Wiederholungen der Sequenz GGGGCC, die bei gesunden Personen weniger als 20-mal vorkommt“, erläutert Prof. Edbauer, der sowohl am DZNE als auch an der LMU tätig ist. „Erstaunlich ist allerdings, dass daraus Proteine entstehen. Denn diese Wiederholungen liegen in einem Bereich der DNA, der eigentlich nicht in Proteine übersetzt wird.“
Vermeintlich stummer Bereich des Erbguts
In der DNA sind die Baupläne für Proteine hinterlegt. Am Anfang eines solchen Bauplans steht nahezu immer ein fest definiertes molekulares Startsignal. Die Region mit den vielfachen Wiederholungen enthält jedoch kein gängiges Startsignal und sollte daher nicht in Proteine übersetzt werden. Offenbar wird die Prozesskette der Proteinsynthese hier anders als sonst üblich in Gang gesetzt. „Man kennt zwei sehr seltene Alternativen zum gängigen Mechanismus. Welche Vorgänge hier wirksam sind, wissen wir bisher nicht“, so Prof. Haass, Standortsprecher des DZNE in München und Leiter des Lehrstuhls für Stoffwechselbiochemie an der LMU.
Durch Experimente in Zellkulturen konnten die Forscher jedoch nachweisen, dass überlange Abfolgen der Sequenz GGGGCC – auch ohne das gängige Startsignal – tatsächlich in Proteine übersetzt werden. Die gleichen Proteine konnten sie auch in den typischen Partikeln finden, die sich im Gehirn von Patienten ansammeln: Es handelt sich dabei um sehr lange „Dipeptid-Repeat Proteine“, die aus einer Verkettung immer gleicher Bausteine bestehen.
„Das sind sehr ungewöhnliche Proteine, die ansonsten im Körper überhaupt nicht vorkommen“, sagt Edbauer. „Soweit wir wissen, sind sie völlig nutzlos und sehr schlecht löslich. Deswegen bilden sie Aggregate und scheinen die Nervenzelle zu schädigen. Der schädigende Einfluss ist noch nicht eindeutig bewiesen, aber vieles spricht dafür.“ Ihre Besonderheit mache diese Proteine jedenfalls zu einem interessanten Ziel für neuartige Therapien. „Da der Mechanismus ihrer Herstellung so ungewöhnlich ist, finden wir vielleicht Wege, ihre Entstehung gezielt zu hemmen, ohne die Synthese anderer Proteine zu stören. Man könnte auch gegen ihre Aggregation vorgehen und versuchen, ihren Abbau zu beschleunigen.“
Ein Patentantrag ist eingereicht und die Wissenschaftler haben große Pläne. „Es ist unser Traum, am DZNE in München eine Therapie für diese schreckliche Krankheiten zu entwickeln“, so Haass und Edbauer.
Originalveröffentlichung
The C9orf72 GGGGCC Repeat is Translated into Aggregating Dipeptide-Repeat Proteins in FTLD/ALS
Kohji Mori, Shih-Ming Weng, Thomas Arzberger, Stephanie May, Kristin Rentzsch, Elisabeth Kremmer, Bettina Schmid, Hans A. Kretzschmar, Marc Cruts, Christine Van Broeckhoven, Christian Haass, Dieter Edbauer, Science Express, doi: 10.1126/science.1232927.