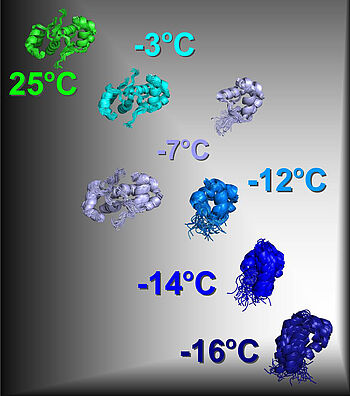
Göttingen, 11. Februar 2013. Wenn Proteine „außer Form“ geraten, können die Folgen fatal sein. Sie verlieren ihre Funktion und in manchen Fällen bilden sie unlösliche, toxische Klumpen, die andere Zellen schädigen und schwere Erkrankungen wie Alzheimer oder Parkinson auslösen können. Forscher des Max-Planck-Instituts für biophysikalische Chemie und des Deutschen Zentrums für Neurodegenerative Erkrankungen in Göttingen haben jetzt mit polnischen Kollegen erstmals in atomarem Detail „gefilmt“, wie sich ein Protein graduell entfaltet. Mit einem kombinierten Einsatz von Kälte und NMR-Spektroskopie gelang es den Wissenschaftlern, sieben Zwischenformen des CylR2-Proteins während der Abkühlung von 25°C auf -16°C sichtbar zu machen. Ihre Ergebnisse zeigen: Die instabilste Zwischenform spielt eine Schlüsselrolle bei der Proteinfaltung. Die Erkenntnisse der Forscher können zu einem verbesserten Verständnis beitragen, wie Proteine ihre Struktur annehmen und sich im Krankheitsfall falsch falten. (Nature Chemical Biology, 10. Februar 2013)
Ob Alzheimer, Parkinson oder Chorea Huntington – alle drei Erkrankungen haben eine gemeinsame Ursache: Falsch gefaltete Proteine lagern sich im Gehirn der Betroffenen zu unlöslichen Klumpen zusammen und zerstören die Nervenzellen. Eine der wichtigsten Fragen der Biowissenschaften und Medizin ist daher, wie Proteine – die Werkzeuge lebender Zellen – ihre dreidimensionale Form erlangen oder verlieren. Denn nur, wenn ihre Ketten von Aminosäuren in einer bestimmten Weise räumlich gefaltet sind, können Proteine ihre Aufgaben erfüllen.
Was genau passiert, wenn Proteine sich falten oder entfalten, konnte man bisher nur schwer untersuchen. Mit Hitze und Druck verliert ein Protein zwar leicht seine Form – und damit seine Funktion. Doch um es bei seinem Entfaltungsprozess direkt zu beobachten, sind die gängigen Methoden nicht geeignet. Seine Zwischenformen bei der Proteinentfaltung sind viel zu kurzlebig.
Mit einem neuen Ansatz ist es Forschern jetzt erstmals gelungen, den komplexen Prozess der Proteinentfaltung zu „filmen“. Wissenschaftler des Max-Planck-Instituts für biophysikalische Chemie (MPIbpc) und des Deutschen Zentrums für Neurodegenerative Erkrankungen (DZNE) am Standort Göttingen haben gemeinsam mit ihren Kollegen an der Polnischen Akademie der Wissenschaften in Warschau und der Universität Warschau in atomarer Auflösung sichtbar gemacht, wie ein Protein schrittweise „außer Form“ gerät. Dabei setzten sie auf Kälte. „Wird ein Protein langsam abgekühlt, reichern sich seine Zwischenstufen in größerer Menge an als bei gängigen Methoden wie Hitze, Druck oder Harnstoff. Unsere Hoffnung war, dass diese Mengen ausreichen, um sie mit der Kernspinresonanz (NMR)-Spektroskopie zu untersuchen“, so Markus Zweckstetter, Leiter der Forschungsgruppen „Proteinstrukturbestimmung mittels NMR“ am MPIbpc und „Strukturbiologie bei demenziellen Erkrankungen“ am DZNE-Standort Göttingen.